MEGA program을 사용한 Phylogenetic tree 작성법
1. 메모장을 사용하여 샘플의 이름 및 sequence를 기입
바른 예)
>Sequence_1
GAATTC---
틀린 예)
>Sequence 1
GAATTC---
※ '>' 부호는 A, T, G, C 가 아닌 일반 텍스트로 인식합니다.
※ 샘플 이름에 공란이 있으면 안됩니다. (띄어쓰기 하면 안됨)
(MEGA 프로그램에서는 공란있어도 가능하지만요)
※ sequence는 대문자, 소문자 무관합니다.
※ 각 샘플들 사이에 줄 띄우기는 있어도, 없어도, 한줄 띄워도, 두줄 띄워도 무관합니다.
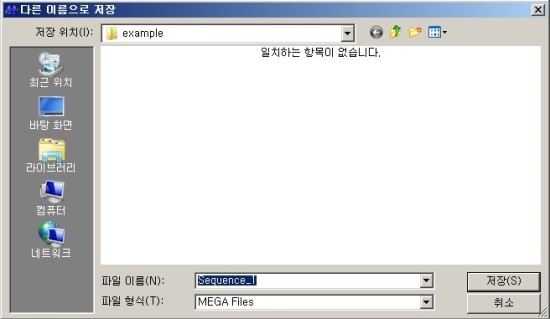
2. 파일 저장
(파일 - 다른이름으로 저장 - 파일형식 : 모든 파일 - 저장할 sequence이름.fas)
예)
일반적인 저장 : Sequence_1.txt
변경해야할 저장 : Sequence_1.fas
틀린 예)
Sequence 1.fas
※ 역시 파일 이름에 공란이 있으면 안됩니다. ( _ 사용하세요)
3. MEGA program 사용
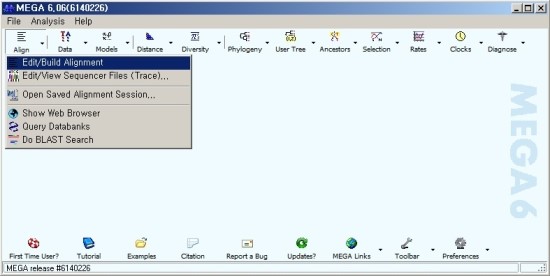
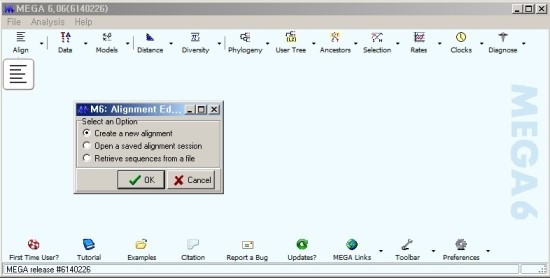
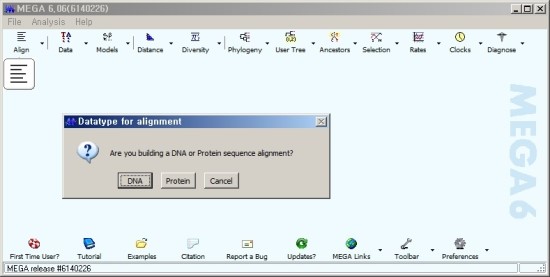
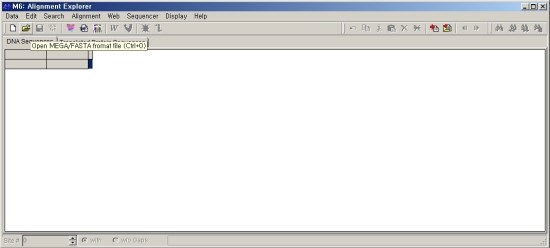
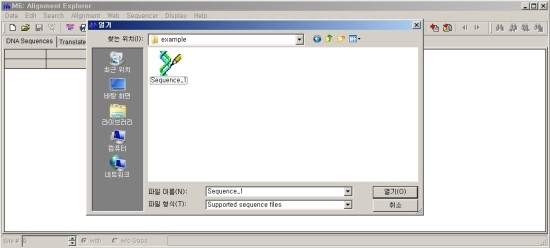
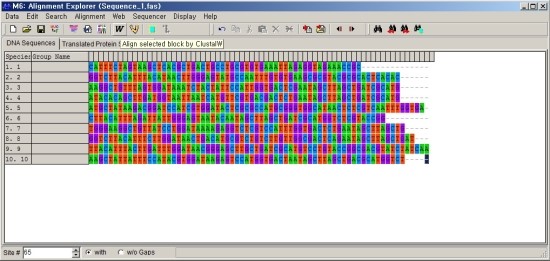
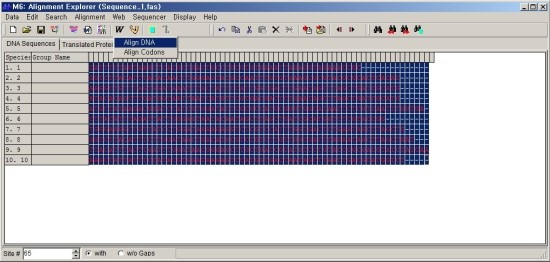
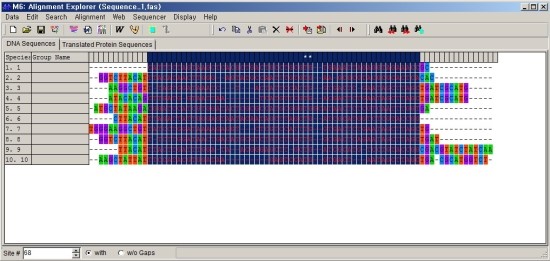
Phylogenetic tree는 일정한 염기서열에서 차이를 확인하는 것이기 때문에
공통된 부분 이외의 부분을 잘라내어야 합니다.
하나라도 - 로 표기된 부분이 없는 부분만 사용하고 나머지는 [delete]키를 눌러 삭제합니다

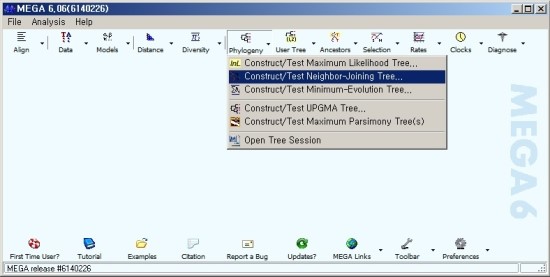
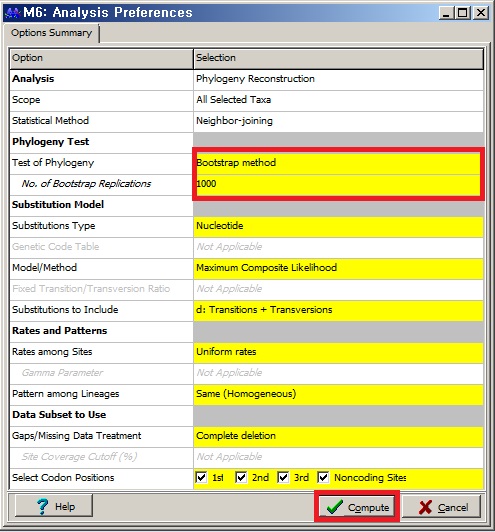
[Test of Phylogeny] - [Bootstrap method] 선택
[No. of Bootstrap Replications] - 1000 기입 (500 해도 상관없음)
(기본으로 500 설정되어 있는데, 이는 몇번 반복 실행을 하여 같은 결과가 나오는지를 나타냅니다.)
이후 [Compute]
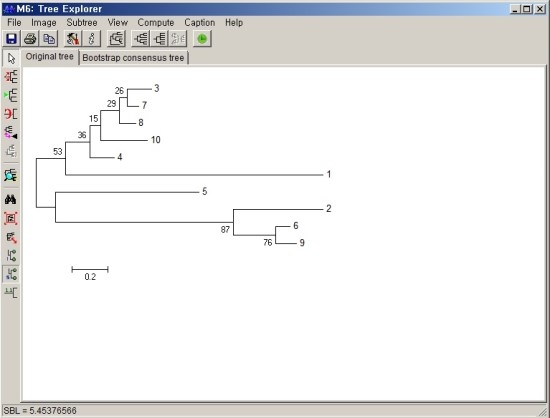
원하는 Phylogenetic tree가 나오게 됩니다.